Contrôle de la croissance par les nutriments
Inserm U1151, Institut Necker Enfants MaladesFaculté de Médecine Paris Descartes, Bat. Leriche, porte 9
14 Rue Maria Helena Vieira Da Silva, CS61431
75993 Paris cedex 14 - Paris
Site web - mario.pende@inserm.fr - 0172606386
Responsable d'équipe
Mario Pende

Thématiques de recherche
Chez les métazoaires, la disponibilité des nutriments et des facteurs de croissance contrôle le nombre de cellules, la taille et l’homéostasie métabolique. Nous étudions les programmes spécifiques sous-jacents à ces réponses et leur coordination par des mécanismes de transduction du signal.
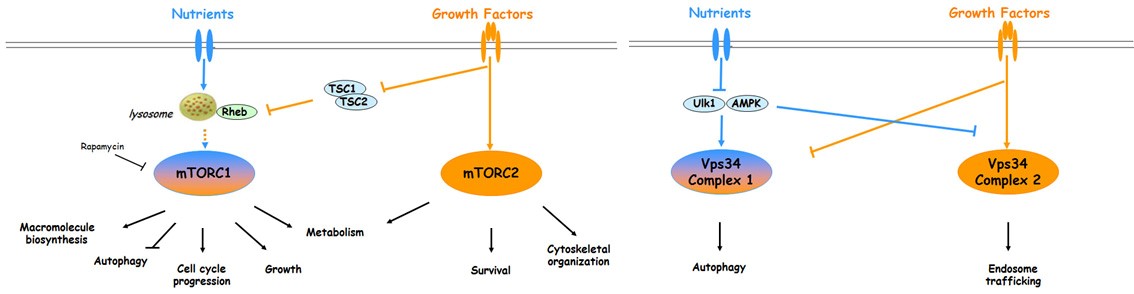
Nous nous intéressons à deux voies de transduction des signaux nutritifs, mTOR (cible mammalienne de la rapamycine) et le complexe Vps15 / Vps34 (Vacuolar Protein Sorting 15/34). Ces anciennes voies sont présentes dans chaque cellule eucaryote, des organismes unicellulaires comme la levure à l’homme. Ils fonctionnent comme des mécanismes essentiels qui déterminent comment la croissance et le métabolisme s’adaptent aux signaux nutritionnels. mTOR est une protéine Ser / Thr kinase, tandis que le complexe Vps15 / Vps34 est une lipide kinase avec une activité phosphatidylinositol 3-kinase (PI3K de classe III). Les mécanismes de transduction déclenchés par mTOR et PI3K de classe III sont complexes. Dans les cellules de mammifères, les deux kinases sont engagées dans de multiples complexes qui ont différentes localisations, cibles et sensibilités aux signaux en amont, comme les nutriments et l’insuline.
Nous avons contribué à démontrer que cette régulation différentielle peut expliquer de nombreuses réponses physiologiques à la nutrition. Par exemple, pourquoi les nutriments et l’insuline sont synergiques pour la croissance cellulaire, bien que les nutriments provoquent une résistance à l’action métabolique de l’insuline. Ou pourquoi l’insuline inhibe l’autophagie, bien qu’elle stimule le trafic des récepteurs.
Au cours des quinze dernières années, nous avons généré et caractérisé un large panel de souris mutantes dans les voies mTOR et PI3K de classe III. Nous avons été impliqués dans la révélation de phénotypes uniques et intéressants qui ont augmenté notre connaissance des rôles de mTOR / PI3K de classe III en physiopathologie: des mutants avec de petites cellules (Pende et al., Nature, 2000, Ohanna et al., Nature Cell Biol, 2005), des mutants de la biogenèse des ribosomes (Fumagalli et al., Nature Cell Biol, 2009), des mutants résistants à la tumorigenèse dans des tissus spécifiques et après des insultes oncogéniques spécifiques (Alliouachene et al., JCI, 2008, Nature Comm. Patitucci et al., JCI, 2017), des mutants avec une maladie autophago-lysosomale (Nemazanyy et al., EMBO Mol Med, 2013), des mutants mimant la restriction calorique et favorisant la longévité (Aguilar et al. Metabolism, 2007, Barilari et al., EMBO J, 2017), des mutants avec une action modifiée de l’insuline (Nemazanyy et al., Nature Comm., 2015, Treins et al., Mol Cel Biol, 2012).
Figure descriptive
Publications
– Patitucci C., Couchy G., Bagattin A., Cañeque T., de Reyniès A., Scoazec J-Y, Rodriguez R., Pontoglio M., Zucman-Rossi J., Pende M.* and Panasyuk G. (2017) *corresponding author. HNF1α suppresses steatosis associated liver cancer by inhibiting PPARγ transcription. Journal of Clinical Investigation, 127, 1873-1888, doi: 10.1172/JCI90327.
– Barilari M., Bonfils G., Treins C., Koka V., De Villeneuve D., Fagrega S., Pende M. (2017) ZRF1 is a novel S6 kinase substrate that drives the senescence program. EMBO J., 36, 736-750. doi: 10.15252/embj.201694966.
– Nemazanyy I., Montagnac G., Russell R.C., Morzyglod L., Burnol A.F., Guan K.L., Pende M. *, Panasyuk G. (2015) *corresponding author .Class III PI3K regulates organismal glucose homeostasis by providing negative feedback on hepatic insulin signalling. Nature Communications, 21, 8283. doi: 10.1038/ncomms9283.
– Liang N., Zhang C., Dill P., Panasyuk G., Pion D., Koka V., Gallazzini M., Olson E.N., Lam H., Henske E.P., Dong Z., Apte U., Pallet N., Johnson R.L., Terzi F., Kwiatkowski D.J., Scoazec J-Y., Martignoni G., Pende M. (2014) Regulation of YAP by mTOR and autophagy reveals a therapeutic target of tuberous sclerosis complex. J Exp Med. 211, 2249-2263. doi: 10.1084/jem.20140341.
– Nemazanyy I., Blaauw B., Paolini C., Caillaud C., Protasi F., Mueller A., Proikas-Cezanne T., Russell R.C., Guan K-L., Nishino I., Sandri M., Pende M.*, Panasyuk G. (2013) *corresponding author. Defects of Vps15 in skeletal muscles lead to autophagic vacuolar myopathy and lysosomal disease. EMBO Mol Med, 5, 870-890. doi: 10.1002/emmm.201202057.
– Chauvin C., Koka V., Nouschi A., Mieulet V., Hoareau-Aveilla C., Dreazen A., Cagnard N., Carpentier W., Kiss T., Meyuhas O., Pende M. (2013) Ribosomal protein S6 kinase activity controls the Ribosome Biogenesis transcriptional program. Oncogene, 33, 474-483. doi: 10.1038/onc.2012.606. Epub 2013 Jan 14.
– Treins C., Alliouachene S., Hassouna R., Xie Y., Birnbaum M.J., Pende M. (2012) The combined deletion of S6K1 and Akt2 deteriorates glycemic control in a high-fat diet. Mol Cell Biol, 32, 4001-4011.
– Panasyuk G., Espeillac C., Chauvin C., Pradelli L.A., Horie Y., Suzuki A., Annicotte J.S., Fajas L., Foretz M., Verdeguer F., Pontoglio M., Ferré P., Scoazec J.Y., Birnbaum M.J., Ricci J.E., Pende M. (2012) PPARγ contributes to PKM2 and HK2 expression in fatty liver. Nature Communications, 3:672. doi: 10.1038/ncomms1667.
– Espeillac C., Mitchell C., Celton-Morizur S., Chauvin C., Koka V., Gillet C., Albrecht J.H., Desdouets C., Pende M. (2011) S6 kinase 1 activity is required for rapamycin-sensitive liver proliferation after mouse hepatectomy. Journal of Clinical Investigation, 121, 2821-2832.
– Alliouachene S., Tuttle R.L., Boumard S., Lapointe T., Berissi S., Germain S., Jaubert F., Tosh D., Birnbaum M.J., Pende M. (2008) Constitutively active Akt1 expression in mouse pancreas requires S6 Kinase 1 for insulinoma formation. Journal of Clinical Investigation, 118, 3629-3638.
– Aguilar V., Alliouachene S., Sotiropoulos A., Sobering A., Athea Y., Djouadi F., Miraux S., Thiaudière E., Foretz M., Viollet B., Diolez P., Bastin J., Benit P., Rustin P., Carling D., Sandri M., Ventura-Clapier R., Pende M. (2007) S6 kinase deletion suppresses muscle growth adaptations to nutrient availability by activating AMP kinase. Cell Metabolism, 5, 476-487.
– Ohanna M., Sobering A.K., Lapointe T., Lorenzo L., Praud C., Petroulakis E., Sonenberg N., Kelly P.A., Sotiropoulos A., Pende M. (2005) Atrophy of S6K1-/- skeletal muscle cells reveals distinct mTOR effectors for cell cycle and size control. Nat. Cell Biol. 7, 286-294.
Composition de l'équipe
Ganna Panasyuk, CR1 Inserm
Anne Sophie Armand, MCU Paris Descartes University
Catherine Caillaud, MCU-PH, Hopital Necker
Muriel Girard, MCU-PH, Hopital Necker
Vonda Koka, Ingenieur IE, Inserm
Delphine de Villeneuve, Ingenieur AI, Paris Descartes University
Martina Bonucci, PhD student
Nicolas Kuperwasser, Postdoc
Talha Rashid, PhD student
Ning Liang, Postdoc
Anton Iershov, Postdoc
Chantal Alkhoury, PhD student,
Paul Crespin, M2 Student
Tiffen El Belaidi, M2 Student
Camille Streiff, M2 Student
Edward Chaloui, M2 Student