Dynamique de la Compartimentation Cellulaire chez les plantes
Institut des Sciences du Végétal , UPR 2355, CNRS.Imagif- Centre de Recherche de Gif sur Yvette, FRC 3115, CNRS.
(I2BC – Institut de Biologie Intégrative de la Cellule au 01/01/2015) - Gif sur Yvette
Site web - bsj@isv.cnrs-gif.fr -
Responsable d'équipe
Béatrice Satiat-Jeunemaitre

Thématiques de recherche
Le groupe contribue à la compréhension des mécanismes régulant la morphogenèse et l’organisation fonctionnelle du système endomembranaire chez les plantes supérieures, et utilisant son expertise en Imagerie cellulaire et moléculaire du trafic endomembranaire. Les récents travaux de l’équipe ont montré l’intérêt d’utiliser la cellule végétale comme modèle expérimental pour rechercher les mécanismes subcellulaires régulant l’autophagie (Le Bars et al., 2014).
La morphologie d’un autophagosome est particulière de par la présence d’une double membrane venant encercler un volume cytoplasmique. Elle diffère donc du profil des compartiments constituant le système endomembranaire de la cellule qui eux, sont délimités par une seule membrane. Cet état de fait questionne le mode de formation des autophagosomes, et ses interactions potentielles avec les autres compartiments. La formation des autophagosomes est associée à une cascade d’évènements moléculaires complexes impliquant une batterie de protéines ATG. La localisation précise de ces évènements in cellulo dans le temps et l’espace reste encore à clarifier. L’équipe s’efforce de comprendre les mécanismes sous-jacents à la formation des autophagosomes dans les cellules végétales, et leurs rôles dans la réponse des plantes aux différents stress imposés par l’environnement. L’imagerie dynamique des proteines ATG par microscopie multiéchelles et multimodales permet d’analyser les interactions moléculaires gouvernant la mise en place des membranes des autophagosomes dans la cellule, infirmant ou confirmant les hypothèses de travail issues de l’étude d’autres modèles expérimentaux ou par d’autres méthodes. L’expertise de l’équipe en imagerie permet aussi des collaborations sur d’autres systèmes modèles (Djeddi et al., 2012 ; Manil-Ségalen et al., 2014).
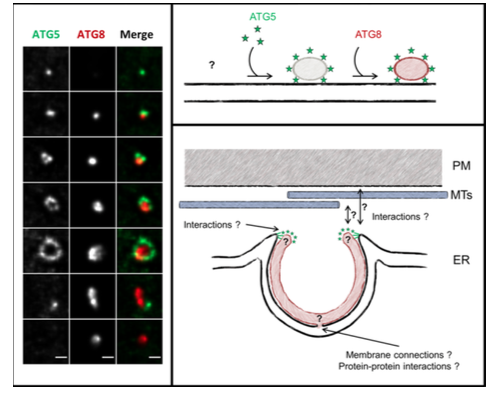
Nous avons récemment démontré, dans des conditions de carences nutritionnelles, une association étroite entre l’autophagosome en formation (le phagophore) et le réticulum endoplasmique sur lequel il semble s’adosser (LeBars et al., 2014a,b).
Notre modèle de formation des autophagosomes soutient l’hypothèse du réticulum endoplasmique comme acteur essentiel. Nous montrons que l’association d’ATG5 avec le réticulum se fait essentiellement sur la partie de la membrane en contact étroit avec le plasmalemme (PM), questionnant l’importance potentielle des sites de contacts membranaires ER/PLM dans la formation d’une structure autophagique. D’autres questions restent à résoudre : quels sont les mécanismes à la base de l’aassociation première entre le phagophore marqué par ATG5 et le réticulum endoplasmique ? Comment le phagophore évolue en une structure courbe ? Existe-t-il une signature moléculaire protéique ou lipidique spécifique sur le réticulum déterminant un « autophagosome formation site » (“ERAFS”) ? Nous abordons aujourd’hui ces questions en utilisant des approches combinées de biologie cellulaire, moléculaire et biochimiques, et en utilisant l’expertise du laboratoire en Biologie Végétale et en Imagerie. Nous serons heureux de partager cet effort de recherche avec d’autres équipes « Autophagie ».
Figure descriptive
Publications
– Le Bars, R., Marion, J., Le Borgne, R., Satiat-Jeunemaitre, B. & Bianchi, MW. (2014) ATG5 defines a phagophore domain connected to the endoplasmic reticulum during autophagosome formation in plants. Nat. Commun. 5, 4121 (2014).
– Djeddi, A., Michelet, X., Culetto, E., Alberti, A., Barois, N. & Legouis, R. (2012) Induction of autophagy in ESCRT mutants is an adaptive response for cell survival in C. elegans. Journal of cell science 125, 685-694.
– Le Bars R., Marion J., Satiat-Jeunemaitre B., Bianchi MW. (2014). Folding into an autophagosome : Atg5 sheds
light on how plants do it. Autophagy 10, 1-3
– Manil-Ségalen M, Lefebvre C, Jenzer C, Trichet M, Boulogne C, Satiat-Jeunemaitre B, Legouis R. (2014) The C.
elegans LC3 acts downstream of GABARAP to degrade autophagosomes by interacting with the HOPS subunit VPS39.Dev Cell. 28(1):43-55 Erratum in: Dev Cell. 2014 Jul 14;30(1):110.
Composition de l'équipe
Groupe « Morphodynamique du système endomembranaire »
Michele Wolfe Bianchi (MCU UPEC) (1)
Romain le Bars (PhD) (2)
Jessica Marion (AI, CNRS) (3)
Frederic Coquelle (MCU Paris-Sud) (4)
Arthur Molines (Etudiant en thèse)
Mebarek Temagoult (AI, CDD) (5)
Groupe “Pôle Imagerie et Biologie Cellulaire d’Imagif”
Claire Boulogne (IR) (6)
Mickael Bourge (IE) (7)
Cynthia Gillet (AI) (8)
Romain LeBars (CDD, IE) (9)
Laetitia Besse (CDD, IE) (10)