Vin-Aliments-Microbiologie-Stress (VALMIS)
UMR PAM, Esplanade Erasme, 21 000, DIJON - DijonSite web - pierre.lapaquette@u-bourgogne.fr -
Responsable d'équipe
Pierre Lapaquette

Thématiques de recherche
L’équipe VALMiS est spécialisée dans l’étude des micro-organismes d’intérêt biotechnologique (fabrication d’aliments, probiotiques pour la santé humaine et animal) et plus particulièrement, leurs interactions avec l’environnement (organisme hôte, autres microorganismes, matrice alimentaire ou vin).
Une première partie de l’équipe s’intéresse aux mécanismes que développent les microorganismes nécessaires à la vinification (bactéries et levures) afin de survivre aux conditions hostiles du milieu vin (comprenant l’acidification, présence d’éthanol, manque de nutriments). Ils étudient également les relations existantes entre les communautés microbiennes présentes dans le vin et les risques d’altérations de celui ci par des microorganismes délétères.
Une seconde partie de l’équipe, alliant des cliniciens, s’intéresse aux bactéries et levures probiotiques. Les probiotiques sont définis comme étant des micro-organismes vivants exerçant une action bénéfique sur la santé de l’hôte qui les ingère en améliorant l’équilibre de la flore intestinale, au-delà des effets nutritionnels traditionnels. Notre équipe étudie en particulier les mécanismes de résistance aux stress de ces microorganismes dans le tractus gastro-intestinal ainsi que les molécules d’intérêts produites par ceux-ci et pouvant interagir avec la réponse immunitaire de l’hôte (immunomodulation) ou le microbiote intestinal. Les effets de ces probiotiques sont testés dans différentes contextes physiopathologiques incluant les maladies inflammatoires chroniques intestinales et les infections (pathogènes intestinaux tel que Salmonella spp et Candida albicans).
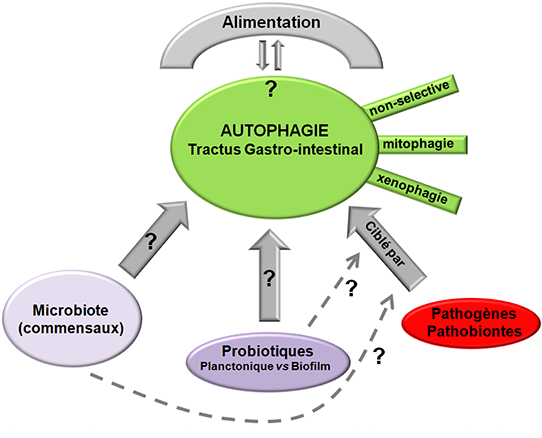
Une thématique émergente de notre équipe est de développer des microorganismes probiotiques innovants, naturels ou génétiquement modifiés, pouvant contribuer à l’homéostasie intestinal, en particulier en stimulant le processus d’autophagie. En effet, à ce jour, il existe un très grand nombre d’études qui ont analysé les effets de microorganismes pathogènes sur l’autophagie, mais très peu de données existent sur la régulation du processus autophagique par les microorganismes commensaux du microbiote intestinal ou des microorganismes probiotiques. Notre projet de recherche a un double objectif : (1) comprendre l’interaction de bactéries commensales et probiotiques, ou du microbiote dans sa globalité, avec le processus d’autophagie au niveau intestinal que ce soit en condition basale (homéostasie) ou lors de situations physiopathologiques stimulant l’autophagie (infections par des micro-organismes pathogènes et maladies inflammatoires chroniques intestinales), et (2) utiliser des bactéries probiotiques naturelles ou génétiquement modifiées pour stimuler l’autophagie et ainsi renforcer l’homéostasie intestinale. Nous caractérisons les effets de ces microorganismes sur le processus d’autophagie in vitro sur des lignées de cellules épithéliales intestinales et in vivo chez la souris (germ free ou conventionnelles) et le poisson zèbre.
Figure descriptive
Publications
– Aoudia N., Rieu A., Briandet R., Deschamps J., Chluba J., Jego G., Garrido C., Guzzo J. (2016). Biofilms of Lactobacillus plantarum and Lactobacillus fermentum: effect on stress responses, antagonistic effects on pathogen growth and immunomodulatory properties. Food Microbiology, 53, 51-59.
– Goyer M, Loiselet A, Bon F, L’Ollivier C, Laue M, Holland G, Bonnin A, Dalle F (2016). Intestinal Cell Tight Junctions Limit Invasion of Candida albicans through Active Penetration and Endocytosis in the Early Stages of the Interaction of the Fungus with the Intestinal Barrier. PLoS One. 2016 Mar 2;11(3):e0149159.
– Albac S, Schmitz A, Lopez-Alayon C, d’Enfert C, Sautour M, Ducreux A, Labruère-Chazal C, Laue M, Holland G, Bonnin A, Dalle F (2016). Candida albicans is able to use M cells as a portal of entry across the intestinal barrier in vitro. Cell Microbiol. 2016 Feb;18(2):195-210.
– Bastard A, Coelho C, Briandet R, Canette A, Gougeon R, Alexandre H, Guzzo J, Weidmann S (2016) Effect of Biofilm Formation by Oenococcus oeni on Malolactic Fermentation and the Release of Aromatic Compounds in Wine. Front Microbiol. 2016 Apr 27;7:613.
– Lapaquette P, Guzzo J, Bretillon L and Bringer MA. Cellular and molecular connections between autophagy and inflammation. Mediators Inflamm. 2015;2015:398483.
– Darsonval M, Msadek T, Alexandre H, Grandvalet C. (2015)The Antisense RNA Approach: a New Application for In Vivo Investigation of the Stress Response of Oenococcus oeni, a Wine-Associated Lactic Acid Bacterium. Appl Environ Microbiol. 2015 Oct 9;82(1):18-26.
– Grangeteau C, Daniel Gerhards, Sébastien Terrat, Samuel Dequiedt, Hervé Alexandre, Michèle Guilloux-Bénatier, Christian von Wallbrunn, Sandrine Rousseaux (2015) FT-IR spectroscopy: a powerful tool for studying the inter- and intraspecific biodiversity of cultivable yeasts isolated from grape must. Journal of Microbiological Methods 121, 50-58.
– Youzhong L, Rousseaux S, Tourdot-Maréchal R, Sadoudi M, Gougeon G, Schmitt-Kopplin P, Alexandre H (2015). Wine microbiome, a dynamic world of microbial interactions. Critical Reviews in Food Science and Nutrition. 2015 Jun 11:0.
– Rieu A, Aoudia N, Jego G, Chluba J, Yousfi N, Briandet R, Deschamps J, Gasquet B, Monedero V, Garrido C, Guzzo J (2014). The biofilm mode of life boosts the anti-inflammatory properties of Lactobacillus. Cell Microbiol. 2014 Dec;16(12):1836-53.
– Maitre M, Weidmann S, Dubois-Brissonnet F, David V, Covès J, Guzzo J (2014). Adaptation of the wine bacterium Oenococcus oeni to ethanol stress: role of the small heat shock protein Lo18 in membrane integrity. Appl Environ Microbiol. 2014 May;80(10):2973-80.
– Maitre M, Weidmann S, Rieu A, Fenel D, Schoehn G, Ebel C, Coves J, Guzzo J (2012). The oligomer plasticity of the small heat-shock protein Lo18 from Oenococcus oeni influences its role in both membrane stabilization and protein protection. Biochem J. 2012 May 15;444(1):97-104.
– Lapaquette P, Bringer MA, Darfeuille-Michaud A (2012). Defects in autophagy favour adherent-invasive Escherichia coli persistence within macrophages leading to increased pro-inflammatory response. Cell Microbiol. 2012 Jun;14(6):791-807.
– Brest P, Lapaquette P, Souidi M, Lebrigand K, Cesaro A, Vouret-Craviari V, Mari B, Barbry P, Mosnier JF, Hébuterne X, Harel-Bellan A, Mograbi B, Darfeuille-Michaud A, Hofman P. A synonymous variant in IRGM alters a binding site for miR-196 and causes deregulation of IRGM-dependent xenophagy in Crohn’s disease. Nat Genet. 2011 Mar;43(3):242-5.
Composition de l'équipe
Jana Al Azzaz (Doctorante 2ème année)
Hervé Alexandre (PU, Directeur Valmis)
Louise Basmacyan (Ass. Hospitalo-Universitaire)
Fabienne Bon (MCU)
Antonio Camara (Doctorant 1er année)
Frédéric Dalle (PU-PH, Chef de service parasitologie/mycologie, CHU Dijon)
Vanessa David-Vaizant (AI)
Amandine Ducreux (Tech)
Antoine Gobert (Doctorant 1er année)
Cosette Grandvalet (MCU)
Jean Guzzo (PU)
Michèle Guilloux-Benatier (MCU)
Corrine Hatier (Secrétaire)
Arnaud Heumann (Doctorant 2ème année)
Pierre Lapaquette (MCU)
Julie Laurent (Tech)
Alicia Loiselet (Doctorante 2ème année)
Cédric Longin (Doctorant 3ème année)
Luc Perepelkine (Ingénieur d’étude)
Clément Petitgonnet (Doctorante 1ère année)
Aurélie Rieu-Guigon (MCU)
Sandrine Rousseaux (MCU)
Raphaëlle Tourdot-Maréchal (MCU)
Marc Sautour (MCU-PH)
Stéphanie Weidmann-Desroches (MCU)